Ajustement de courbe
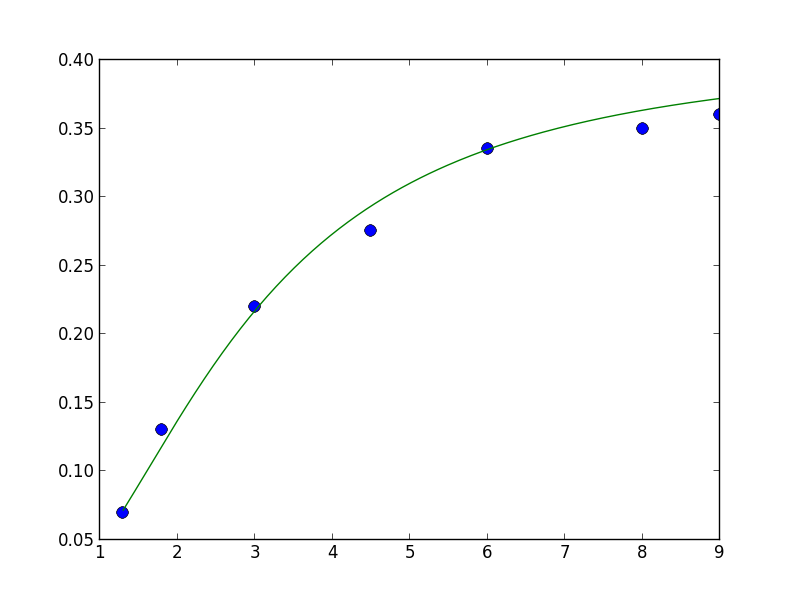
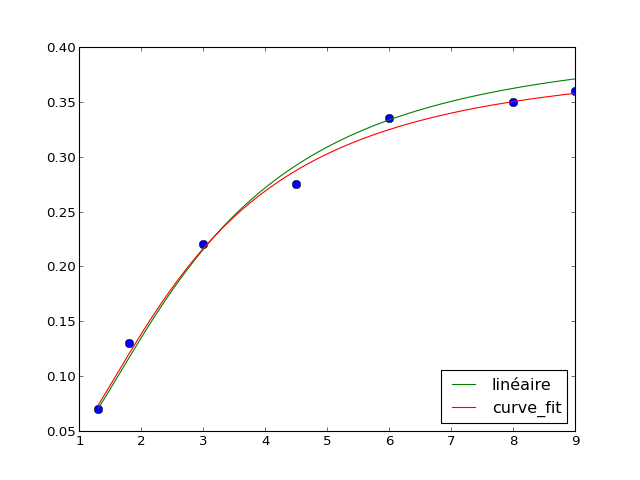
Pour des données expérimentales ou non, on cherche à ajuster des paramètres d'une classe de fonctions définies pour approcher au mieux la courbe expérimentale. Bien souvent la classe de fonction est l'ensemble des polynômes (de degré maximum fixé), de polynômes trigonométrique et on tombe sur la classique régression linéaire. Le "au mieux" dépend aussi de la façon dont on mesure la distance entre courbe expérimentale et classe de fonctions.